登录35斗
中国农科院深圳农业基因组研究所/南京农业大学成功开发新的低成本基因分型技术FBI-seq
全基因组基因型检测是现代育种过程中的重要工具。目前,虽然有多种技术可以进行全基因组基因型检测,但是,昂贵的价格限制了这些技术的广泛使用。尤其是对于非主粮作物的全基因组基因型检测,现有的DNA芯片等技术需要设计、制造芯片等前期成本投入,导致众多非主粮作物仍然缺乏全基因组基因型检测的有效方法。因此,开发低成本且物种通用的全基因组基因型检测技术,可以促进作物基因组选择育种的广泛应用,对我国种业振兴有重要的价值。
JIPB近日在线发表了中国农科院农业基因组研究所常玉晓课题组、钱前院士团队和南京农业大学智海剑教授团队题为“A prolific and robust whole-genome genotyping method using PCR amplification via primer-template mismatched annealing”的方法学论文。该研究前期偶然发现,PCR扩增过程中的非特异性扩增并非杂乱无章,而是有其产生的规律;在此基础上,作者通过增强而非抑制非特异性PCR扩增,开发了一种全新的低成本全基因组基因型检测方法FBI-seq(Foreground and Background Integrated genotyping by sequencing)。对于任意物种,FBI-seq只需要设计一条引物,通过引物与DNA模板间完美匹配(Primer-Template Perfect Annealing, PTPA)的扩增,检测前景基因位点;通过引物与DNA模板间不完美匹配(Primer-Template Mismatched Annealing, PTMA)的扩增,检测基因组上数万个背景位点的基因型。与现有技术相比,FBI-seq实验流程简单,无需前期芯片设计、制造等投入,在基因组选择育种、尤其是非主粮作物的基因组选择育种方面有重要的应用前景。
在本项目开展的初期,为了改进传统的转座子显示技术(Transposon display)用于全基因组基因型检测,作者利用Tn5转座酶替代传统的限制性内切酶和T4 DNA连接酶来打断基因组DNA并连接人工合成的DNA接头(Adaptor),随后利用水稻Rim2转座子特异引物(Specific primer, SP)进行PCR扩增及建库测序。作者意外发现,如果不利用嵌套引物(Nested primer),除了预期的PTPA扩增外,还存在着数以万计的PTMA扩增。
除了转座子引物外,为了验证FBI-seq技术对不同前景基因的检测效果,作者还选择了水稻中稻瘟病抗性基因Pi2和控制粒长的基因qGL3等共6个前景基因,分别设计特异引物进行FBI-seq文库构建及测序,数据分析发现通过引物的PTPA扩增可准确检测这些前景基因位点,同时基于全基因组范围内的PTMA扩增也获得了数以万计的背景基因位点,因此FBI-seq中的PTPA和PTMA扩增特性可成功应用于任意不同前景基因引物。随后,作者又分析了多个水稻品种的不同技术重复间可重复鉴定到的背景位点数量和分布,以及不同抽样数据量下PTMA位点数目的变化特征,进一步验证了PTMA扩增的稳定性和可重复性。
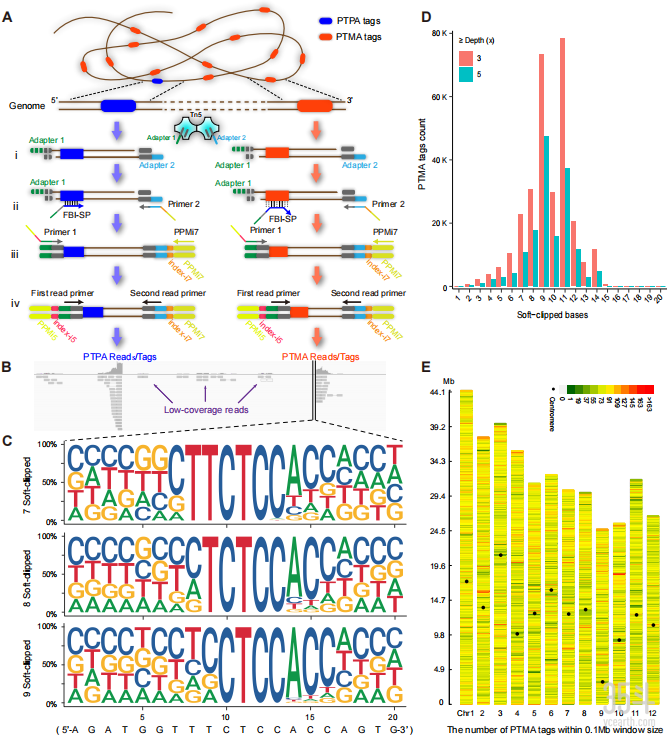
图1. FBI-seq对水稻育种群体进行前景基因和遗传背景检测
为了探究FBI-seq在育种中的实际应用效果,作者选择了一个水稻BC2F4群体进行验证。利用结合在稻瘟病抗性基因Pi2位点的SP引物,对随机选择的5个单株进行FBI-seq文库构建及测序,根据read覆盖深度获得了三类SNP,基于三类SNP分别获得的binmap图谱显示,5个测试单株均直接检测到了Pi2基因位点,遗传背景回复率在83.8~93.0%之间。
既然PTMA的扩增只需要8个左右碱基序列的匹配,因此,作者推测,任意一条引物在不同物种中均可使用。实验也证实,根据水稻基因组设计的多个SP引物在西红柿和豇豆基因组中通过PTMA扩增,也能对其数万个背景位点进行基因型检测,且这些位点在全基因组上均匀分布,说明FBI-seq在其它物种中也有很好的基因型检测能力。
FBI-seq的成功开发,不仅实现了作物育种中对前景基因和遗传背景的同时检测,节省了育种成本,提高了育种效率;同时,作为一种新的全基因组基因型检测技术,FBI-seq也有望在各种作物的真假品种鉴别、指纹图谱检测、种质资源鉴定、遗传图谱构建等领域中发挥重要作用。中国农科院深圳农业基因组所常玉晓研究员、钱前院士和南京农业大学智海剑教授为本论文通讯作者。本项目得到了国家自然科学基金、江苏省现代作物生产协同创新中心等项目的资助。
注:文中如果涉及35斗记者采访的数据,均由受访者提供并确认。
声明:35斗所刊载内容之知识产权为35斗及相关权利人专属所有或持有。转载请联系gao.kp@vcbeat.net。
用户
反馈





